ملف:Tiger phylogenetic relationships.png
من موسوعة المزرعة
اذهب إلى التنقل
اذهب إلى البحث
حجم هذه المعاينة: 574 × 600 بكسل. الأبعاد الأخرى: 230 × 240 بكسل | 460 × 480 بكسل | 735 × 768 بكسل | 980 × 1٬024 بكسل | 2٬001 × 2٬090 بكسل.
الملف الأصلي (2٬001 × 2٬090 بكسل حجم الملف: 684 كيلوبايت، نوع MIME: image/png)
هذا الملف من ويكيميديا كومنز ويمكن استخدامه بواسطة المشاريع الأخرى. الوصف على صفحة وصف الملف هناك معروض بالأسفل.
ملخص
| الوصفTiger phylogenetic relationships.png |
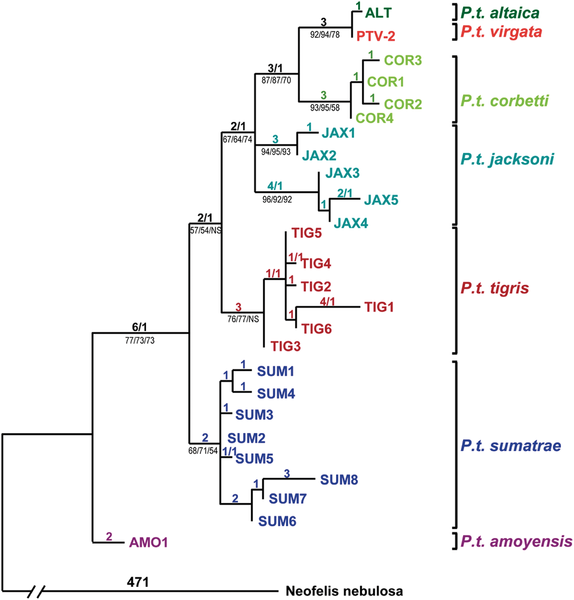
English: Haplotype designations are color coded by subspecies of the tigers that carried them. PTV-2 is a Caspian tiger (Panthera tigris virgata) specimen for which all gene segments attempted (1257 bp) in Caspian tigers were successfully sequenced. Other Caspian tigers produced partial sequences identical to PTV-2. The only exceptions were three individuals, each displaying a single derived nucleotide difference when compared to PTV-2 (found only in that individual and in no other tigers of any subspecies). Likewise, the only mtDNA haplotype carried by Amur or “Siberian” tigers (P. t. altaica) proved to be a single derived step away from the haplotype of PTV-2, suggesting a close relationship between the Amur and Caspian tiger subspecies. Tiger haplotypes carried by all but the Caspian subspecies are from a previously published dataset, while a clouded leopard (Neofelis nebulosa) sequence (GenBank DQ257669) was used to root the tree. The tree depicted was inferred using maximum parsimony, with the number of steps/homoplasies listed above the branches, while (for major clades) bootstrap percentages are listed below branches for maximum parsimony, maximum likelihood and Neighbour Joining methods. We used full length mtDNA sequences of clouded leopard, leopard and snow leopard to root the tree; all combinations of 1, 2 or 3 outgroups yielded trees with similar topology to the one depicted, with the same basal position for the P. t. amoyensis AMO1 haplotype, and a close relationship between P.t. virgata and P. t. altaica haplotypes. doi:info:doi/10.1371/journal.pone.0004125.g002 |
||
| التاريخ | |||
| المصدر | Driscoll CA, Yamaguchi N, Bar-Gal GK, Roca AL, Luo S, et al. (2009) Mitochondrial Phylogeography Illuminates the Origin of the Extinct Caspian Tiger and Its Relationship to the Amur Tiger. PLoS ONE 4(1): e4125. doi:10.1371/journal.pone.0004125 | ||
| المؤلف | Driscoll CA, Yamaguchi N, Bar-Gal GK, Roca AL, Luo S, et al. | ||
| الترخيص (إعادة استخدام هذا الملف) |
|
الشروحات
أضف شرحاً من سطر واحد لما يُمثِّله هذا الملف
العناصر المصورة في هذا الملف
يُصوِّر
١٤ يناير 2009
٧٠٠٬٥٨٦ بايت
٢٬٠٩٠ بكسل
٢٬٠٠١ بكسل
image/png
19f07b91d4e2594a77d4f1b6c393fb9bd37184f0
تاريخ الملف
اضغط على زمن/تاريخ لرؤية الملف كما بدا في هذا الزمن.
| زمن/تاريخ | صورة مصغرة | الأبعاد | مستخدم | تعليق | |
|---|---|---|---|---|---|
| حالي | 10:41، 26 فبراير 2012 |  | 2٬001 × 2٬090 (684 كيلوبايت) | Pmaas |
استخدام الملف
الصفحة التالية تستخدم هذا الملف:
بيانات ميتا
هذا الملف يحتوي على معلومات إضافية، غالبا ما تكون أضيفت من قبل الكاميرا الرقمية أو الماسح الضوئي المستخدم في إنشاء الملف.
إذا كان الملف قد عدل عن حالته الأصلية، فبعض التفاصيل قد لا تعبر عن الملف المعدل.
| المؤلف |
|
|---|---|
| الدقة الأفقية | 118٫11 دي بي سي |
| الدقة الرأسية | 118٫11 دي بي سي |
| البرمجيات المستخدمة |
|
